Tạp chí Frontiers in Molecular Biosciences công bố đề tài nghiên cứu của Gene Solutions “Lượng giá xét nghiệm và đánh giá ứng dụng lâm sàng của K-4CARE™: xét nghiệm khảo sát toàn diện các chỉ dấu phân tử khối u (CGP) kết hợp với theo dõi tồn dư khối u được cá thể hóa (MRD)”
Bối cảnh: Xét nghiệm dấu ấn sinh học đã dần trở thành tiêu chuẩn trong chuyên ngành ung thư, giúp bác sĩ đưa ra những lựa chọn tối ưu cho người bệnh. So với xét nghiệm đơn gen hoặc những panel với số lượng gen giới hạn, xét nghiệm phân tích toàn diện các chỉ dấu phân tử khối u (CGP) được đánh giá vượt trội hơn; bởi phương pháp này giúp tiết kiệm thời gian và mẫu mô cũng như mang lại hiệu quả phân tích cao. Đề tài được tạp chí Frontiers công bố lần này là nghiên cứu nhằm lượng giá phân tích chuyên sâu về K-4CARE – một xét nghiệm CGP tích hợp theo dõi DNA khối u tự do lưu hành trong máu (ctDNA) để truy tìm tồn dư giúp đánh giá điều trị và tiên lượng tái phát ung thư.
Phương pháp: Nghiên cứu đã lượng giá tính ứng dụng của K-4CARE – xét nghiệm khảo sát toàn diện các chỉ dấu phân tử khối u (CGP) kết hợp theo dõi tồn dư của khối u. Bằng cách sử dụng sử dụng một panel gồm 473 gen liên quan đến ung thư với tổng chiều dài khảo sát là 1,7 Mb. Chuẩn tham chiếu đã được sử dụng để đánh giá ngưỡng phát hiện (LOD), độ tương đồng, độ nhạy, độ đặc hiệu và độ chính xác của xét nghiệm để phát hiện biến thể đơn nucleotide (SNV), chèn/ xóa đoạn nhỏ (Indels), khuếch đại gen và kết hợp gen, mất ổn định vi vệ tinh (MSI) và gánh nặng đột biến khối u (TMB).
Nghiên cứu sau đó được so sánh với các phương pháp khác bằng cách sử dụng 155 mẫu lâm sàng từ 10 loại ung thư. Trong một số bệnh ung thư được chọn lọc, những đột biến từ mô u, được xếp hạng theo thuật toán độc quyền của Gene Solutions và được sử dụng để phát hiện ctDNA trong huyết tương.
Kết quả: Để phát hiện SNV và Indels, kết hợp và khuếch đại gen, xét nghiệm có độ nhạy lần lượt là >99%, 94% và >99%, và độ đặc hiệu >99%. Việc phát hiện các biến thể dòng mầm cũng đạt được độ nhạy và độ đặc hiệu >99%. Đối với gánh nặng đột biến TMB, hệ số tương quan giữa giải trình tự toàn bộ bộ gen và giải trình tự 473 gen mục tiêu của Gene Solutions là 97%. Phân tích MSI khi được so sánh với phương pháp phản ứng chuỗi polymerase (PCR-MSI) cho thấy độ nhạy 94% và độ đặc hiệu >99%. Sự phù hợp giữa xét nghiệm của Gene Solutions và xét nghiệm TruSight Oncology 500 để phát hiện các biến thể khối u, TMB và MSI lần lượt là 100%, 89% và 98%. Khi các đột biến phát hiện từ khối u được sử dụng để theo dõi ctDNA cá thể hóa trong máu (sinh thiết lỏng), tỷ lệ phát hiện ctDNA trong máu là 79% và ứng dụng lâm sàng trong việc theo dõi ung thư được chứng minh trong 2 ca lâm sàng.
Kết luận: Xét nghiệm K-4CARE™ cung cấp thông tin di truyền toàn diện và đáng tin cậy cho cả mục tiêu điều trị đích và liệu pháp miễn dịch. Tích hợp thêm theo dõi ctDNA trong máu các chỉ dấu này giúp các bác sĩ lâm sàng thêm công cụ để theo dõi thêm đáp ứng điều trị và cuối cùng là cung cấp dịch vụ chăm sóc toàn diện cho bệnh nhân ung thư.
Giới thiệu
Liệu pháp điều trị đích được Cơ quan Quản lý Thực phẩm và Dược phẩm Hoa Kỳ (FDA) chấp thuận lần đầu vào năm 2001 (Waarts và cộng sự, 2022). Từ đây, nhiều phát triển đột phá đã làm thay đổi cách điều trị và chẩn đoán ung thư. Những thay đổi này gắn liền với sự tiến bộ trong công nghệ giải trình tự gen, đã cung cấp bức tranh tổng thể của các đột biến trong ung thư và từ đó xác định các đột biến có thể can thiệp được (Waarts và cộng sự, 2022). Khi liệu pháp điều trị đích được chấp thuận, xác định bệnh nhân có đột biến trong bộ gen (genome) để dự đoán đáp ứng với điều trị là rất quan trọng. Do đó, xét nghiệm biomarker được khuyến nghị là tiêu chuẩn chăm sóc để tăng cường y học chính xác trong điều trị ung thư. Khi số lượng các đích điều trị tăng lên, danh sách dấu ấn sinh học (biomarker) cũng được mở rộng, không chỉ về số lượng mà còn về mức độ phức tạp. Bên cạnh việc phát hiện ra các biến thể trong bộ gen đơn lẻ, hiện nay đã xuất hiện thêm một số biomarker có khả năng tìm ra những bộ gen phức tạp hơn, như gánh nặng đột biến khối u (TMB) và mất ổn định vi vệ tinh (MSI). Đối với việc dùng thuốc ức chế chốt kiểm soát miễn dịch (ICIs) trong ung thư giai đoạn tiến xa, TMB và MSI đều đã được FDA chấp thuận là chỉ dấu dự đoán độc lập trong nhiều loại u đặc, bên cạnh phối tử của phân tử chết theo chương trình (PD-L1) (Buttner và cộng sự, 2019).
Trong thực tế lâm sàng, xét nghiệm dấu ấn sinh học (biomarker) thường được thực hiện là các xét nghiệm đơn gen, hotspot hoặc panel với số lượng gen giới hạn do chi phí thấp, quy trình đơn giản và thời gian trả kết quả nhanh. Đặc biệt ở các nước đang phát triển, phương pháp phổ biến dựa trên phản ứng chuỗi polymerase (PCR) như digital droplet PCR để phát hiện các đột biến điểm đơn lẻ; hoặc hóa mô miễn dịch (IHC) và lai huỳnh quang tại chỗ (Fluorescence in situ hybridization – FISH) để phát hiện sự khuếch đại hoặc tái sắp xếp lại gen (Pennell và cộng sự, 2019; Radich và cộng sự, 2022). Giải trình tự gen thế hệ mới (Next-Generation Sequencing – NGS) ngày càng phổ biến nhưng vẫn chỉ tập trung giới hạn cho các bảng gen đã chọn trước. Nhược điểm chính của phương pháp này là không cung cấp đầy đủ tất cả các dấu ấn sinh học (biomarker) đi kèm và không phân tích được những chỉ dấu phức tạp như TMB và MSI, điều này có thể dẫn đến việc bệnh nhân bỏ lỡ cơ hội điều trị. Ví dụ, có báo cáo rằng đối với ung thư biểu mô phổi, tỷ lệ xét nghiệm EGFR đạt con số rất cao (>80%) trong khi tỷ lệ xét nghiệm ALK và ROS1 thấp hơn đáng kể (Pennell và cộng sự, 2019), mặc dù thực tế là đột biến ALK và ROS1 có thể xảy ra trong 10% trường hợp (Song và cộng sự, 2017; Đặng và cộng sự, 2020). Do đó, xét nghiệm khảo sát toàn diện các chỉ dấu phân tử khối u (CGP) giúp phân tích hàng trăm gen trong một xét nghiệm duy nhất sẽ là một giải pháp hiệu quả hơn. CGP có thể tổng hợp kết quả của nhiều dấu ấn sinh học cho cả liệu pháp đã được phê duyệt và liệu pháp mới mà vẫn bảo tồn các mẫu mô sinh thiết quý giá. Việc biết trước các phương án điều trị có thể giúp bác sĩ đưa ra giải pháp điều trị tối ưu hoặc thay đổi liệu pháp kịp thời cho bệnh nhân.
Vấn đề được quan tâm nhiều nhất của CGP là chi phí, yêu cầu thiết bị chuyên dụng và kỹ thuật viên phải có chuyên môn cao. Do đó, xét nghiệm CGP thường sẽ không có sẵn hoặc không phải là giải pháp khả thi về mặt tài chính cho phần lớn bệnh nhân ở các quốc gia có nguồn lực hạn chế, vì vậy mà CGP vẫn là đang một thách thức lớn ở nhiều quốc gia. Hơn nữa, chất lượng của các mô sinh thiết khối u (FFPE) cần được cố định bằng formalin và đúc parafin. DNA và đặc biệt là RNA có thể bị phân hủy nặng nề nếu không có điều kiện cố định và bảo quản chất lượng. Do đó, hạn chế về cả số lượng và chất lượng mẫu đòi hỏi phải được tối ưu hóa. Các mẫu FFPE có cấu trúc mô không còn nguyên vẹn, không chất lượng có thể không sử dụng được. Hơn nữa, CGP cũng từng nhận đánh giá tiêu cực vì phát hiện nhiều đột biến không có ý nghĩa điều trị (Christofyllakis và cộng sự, 2022). Nhưng nếu xét nghiệm đột biến này được tận dụng để phát hiện theo dõi DNA khối u tự do lưu hành trong máu (ctDNA) sẽ tạo điều kiện giám sát gánh nặng đột biến khối u và đáp ứng điều trị của bệnh nhân theo thời gian thực. Lợi ích lâm sàng có ý nghĩa của việc giám sát ctDNA để phát hiện tồn dư tối thiểu (MRD) đã được ghi nhận một cách nhất quán (Chin và cộng sự, 2019).
Trong nghiên cứu này, Gene Solutions đã thiết lập K-4CARE™ (K4C) – xét nghiệm CGP dựa trên mẫu mô sử dụng một panel gồm 473 gen liên quan đến ung thư để phát hiện biến thể đơn nucleotide (SNV), chèn/ xóa đoạn nhỏ (Indels), khuếch đại gen và kết hợp gen, cũng như đánh giá TMB và MSI. Xét nghiệm đã được thẩm định kỹ lưỡng bằng các tiêu chuẩn tham chiếu và tối ưu hóa cho các mẫu lâm sàng của bệnh nhân ung thư tại Việt Nam. Đối với một số loại ung thư nhất định, xét nghiệm K4C cũng tích hợp tính năng phát hiện ctDNA trong sinh thiết lỏng để theo dõi ung thư.
Thu thập mẫu và phương pháp
Thu thập mẫu
Genomic DNA từ mẫu khối u được cố định bằng formalin và đúc parafin (FFPE) cùng với mẫu bạch cầu (WBC) tương ứng của bệnh nhân ung thư được lấy từ Viện Di truyền Y học, thành phố Hồ Chí Minh, Việt Nam. Tất cả các mẫu mô đều được ẩn danh và lưu trữ dưới 2 năm. Tổng cộng có 282 mẫu FFPE từ hơn 10 loại ung thư trong nghiên cứu. Danh sách phân bổ mẫu các thí nghiệm khác nhau được hiển thị trong Bảng Phụ lục S1. Danh sách các tiêu chuẩn tham chiếu thương mại được sử dụng để đánh giá năng lực của xét nghiệm K4C hiển thị trong Bảng Phụ lục S2. Đối với việc phát hiện ctDNA, mẫu huyết tương của 40 bệnh nhân mắc bệnh ung thư đại trực tràng (CRC) hoặc ung thư vú cũng được phân tích trong nghiên cứu (Bảng Phụ lục S1).
Chuẩn bị thư viện và giải trình tự cho FFPE và WBC
Genomic DNA được tách chiết từ mẫu FFPE và WBC đã được mô tả trước đây [(Dang và cộng sự, 2020; Nguyen Hoang và cộng sự, 2023; Nguyen và cộng sự, 2022; Tran và cộng sự, 2021)]. Việc phân mảnh DNA và chuẩn bị thư viện cho các mẫu FFPE và WBC tương ứng thực hiện bằng bộ dụng cụ NEBNext Ultra II FS DNA library prep kit (New England Biolabs, Mỹ) theo hướng dẫn của nhà sản xuất. Thư viện gộp lại với nhau và lai hóa với các đoạn mồi được thiết kế sẵn cho 473 gen liên quan đến ung thư (Integrated DNA Technologies, Mỹ). Panel gen này được chọn lọc từ các cơ sở dữ liệu ung thư công khai, các nghiên cứu hệ gen uy tín và đặc biệt là cơ sở dữ liệu nội bộ từ hơn 10,000 bệnh nhân ung thư. Panel bao gồm toàn bộ exon của 473 gen, vùng promoter của TERT, và và các vùng intron chọn lọc từ 6 gen hay bị dung nạp thường gặp (Bảng Phụ lục S3). Giải trình tự gen song song của thư viện DNA được thực hiện trên máy DNBSEQ-G400 (MGI, Trung Quốc) với độ sâu trung bình 500X cho mẫu FFPE và 500X cho mẫu WBC.
Trong thí nghiệm để xác minh TMB, thư viện DNA cũng được lai hóa với xGen Exome Hyb Panel v2 (IDT, Mỹ) bao phủ toàn bộ exome của bộ gen người. Giải trình tự song song được thực hiện trên máy DNBSEQ-G400 (MGI, Trung Quốc), với độ phủ sâu trung bình cho mẫu FFPE và WBC lần lượt là 250X và 175X. Trong các thí nghiệm so sánh xét nghiệm K4C với TruSight Oncology 500 (Illumina, Mỹ), việc chuẩn bị thư viện và làm giàu DNA mục tiêu cho mẫu FFPE được thực hiện theo hướng dẫn của nhà sản xuất. Thư viện DNA được giải trình tự trên máy NextSeq 550 (Illumina, Mỹ) với giải trình tự hai đầu độ dài 150 base-pair, với độ sâu trung bình 1000X.
Phân tích Bioinformatics
Để kiểm soát chất lượng, Gene Solutions đã sử dụng Illumina DRAGENTM Bio-IT Platform (v3.10) để loại bỏ các dữ liệu và trình tự adapter chất lượng thấp trong các tệp fastq, đồng thời xóa các lần đọc trùng lặp dữ liệu khỏi tệp bam. Mẫu FFPE đạt được chất lượng (QC) của xét nghiệm K4C phải có độ phủ sâu trung bình lớn hơn hoặc bằng 150X và phần trăm phủ ở 100X vượt quá hoặc bằng 75%. Tỷ lệ thất bại là 10.4% và các mẫu không đạt QC đã được loại bỏ khỏi phân tích.
Các đột biến soma được phát hiện từ các tệp bam bởi ống dẫn DRAGENTM Somatic, sử dụng các cặp tumor-normal. Để giảm số lượng artefact liên quan đến xử lý formalin và mẫu mô, bộ lọc sau khi gọi somatic sẽ được bỏ qua soft-clipped và loại bỏ các sự kiện cụm. VEP (phiên bản 105) (McLaren và cộng sự, 2016) được sử dụng để dự đoán tác động của các đột biến và chú thích chúng theo dbSNP (Sherry và cộng sự, 2001), ClinVar (Landrum và cộng sự, 2016), và COSMIC (Tate và cộng sự, 2019). Đối với khuếch đại gen, ống dẫn DRAGENTM Somatic CNV Calling đã được sử dụng. 90 mẫu WBC được sử dụng để xây dựng cơ sở tham chiếu, từ đó tính toán giá trị tỷ lệ sao chép chuẩn hóa. Mức tăng hoặc giảm của các gen mục tiêu được gọi là, với phân khúc = 100 bp. Sự khuếch đại gen được phát hiện khi tỷ lệ sao chép của đoạn trung bình ≥2.0. Để hợp nhất gen, các tệp bam được căn chỉnh từ DRAGENTM với các phần đọc được cắt bớt bởi soft-clipped và đánh dấu đã được phân tích bằng Factera (v1.4.4) (Newman và cộng sự, 2014) với các tham số mặc định. Sự kết hợp được phát hiện khi báo cáo tối thiểu 2 lần đọc kéo dài điểm dừng. Sau đó, cơ sở dữ liệu OncoKB (Chakravarty và cộng sự, 2017) (cập nhật lần cuối vào ngày 02/10/2023) sẽ xác định các thay đổi có đủ điều kiện cho các loại thuốc được FDA phê duyệt (cấp 1–2).
Các đột biến Germline được gọi từ các tệp bam của mẫu WBC bởi ống dẫn DRAGENTM Germline. Các đột biến sau đó được phân loại theo hướng dẫn của Học viện Y học Di truyền và Genomics Mỹ (ACMG) như đã mô tả trước đó [(Tran và cộng sự, 2021; Nguyen và cộng sự, 2023)]. Lúc này, các biến thể gây bệnh và có khả năng gây bệnh cao sẽ được báo cáo.
Việc tính toán TMB được thực hiện bằng cách sử dụng tập lệnh được phát triển bởi Gene Solutions để chia tổng số biến thể thể soma đủ điều kiện cho kích thước của bảng được khảo sát. Một đột biến soma đủ điều kiện phải đáp ứng tất cả các tiêu chí sau: (1) vượt qua các tham số lọc của ống dẫn gọi đột biến, (2) không phải là đột biến Germline và được lọc bởi cơ sở dữ liệu dbSNP (Sherry và cộng sự, 2001), (3) nằm trong vùng mã hóa, (4) không phải là đột biến đồng nghĩa, (5) có VAF ≥5%, độ sâu alen ≥ 5X, tổng độ sâu ≥15X. Kích thước bảng được tính cho các cơ sở trong vùng mã hóa với tổng độ sâu tối thiểu ≥15X. Ngưỡng TMB-Cao (TMB-H) được xác định là 10 đột biến/Mb. Đối với MSI, các vị trí microsatellite không ổn định được phát hiện bởi MSIsensor-pro (v1.2.0) (Jia và cộng sự, 2020) trong chế độ phù hợp với tumor-normal. Nếu tỷ lệ không ổn định trong microsatellite được phát hiện ít nhất 20%, mẫu sẽ được xác định là MSI-Cao (MSI-H).
Đối với các mẫu được phân tích bằng bộ TruSight Oncology 500, quy trình phân tích TSO500 sẽ được thực thi bằng phần mềm DRAGENTM TSO500 (v2.1) trên Nền tảng CNTT sinh học Illumina DRAGENTM (v3.10). Một mẫu (1/40) không đạt chỉ số QC khuyến nghị theo hướng dẫn của nhà sản xuất TSO500 đã bị loại khỏi phân tích.
Phân tích MSI bằng PCR
DNA từ các mẫu FFPE và WBC tiến hành phản ứng PCR đa mồi phát huỳnh quang bằng Hệ thống phân tích MSI, Phiên bản 1.2 (Promega, Hoa Kỳ) để kiểm tra 5 dấu lặp lại đơn nucleotide: BAT-25, BAT-26, MONO-27, NR-21 và NR-24. Các mẫu được chạy trên máy phân tích di truyền SeqStudioTM (ThermoFisher, Mỹ). Những mẫu có ít nhất 2 dấu hiệu thay đổi được phân loại là MSI-H, trong khi các mẫu khác được phân loại là không có bất ổn vi vệ tinh hay vi vệ tinh ổn định (MSS).
Phát hiện ctDNA trong huyết tương
Tất cả các đột biến xuất phát từ FFPE được xếp hạng bằng cách sử dụng thuật toán độc quyền của Gene Solutions như được mô tả trước đây (Nguyen và cộng sự, 2022; Nguyen Hoang và cộng sự, 2023). Các tiêu chí chính bao gồm tần suất alen đột biến (VAF) trong FFPE,các biến thể đột biến gây bệnh dựa vào những dữ liệu ung thư sẵn có cũng như những phân tích nội bộ.Các đột biến được xếp hạng cao nhất (đột biến này đặc trưng cho mỗi bệnh nhân) sẽ được sử dụng để theo dõi ctDNA trong các mẫu huyết tương.
cfDNA đã được trích xuất từ các mẫu huyết tương như đã được mô tả trước đây (Nguyen và cộng sự, 2022; Nguyen Hoang và cộng sự, 2023). Lượng cfDNA tối thiểu là ≥0.15 ng/uL hoặc tổng lượng ≥3 ng. Phân mảnh cfDNA với các vị trí đột biến chọn lọc được khuếch đại bằng KAPA HiFi DNA Polymerase (Roche, USA) và đoạn mồi mục tiêu (PhuSa Biochem, Vietnam). Các đoạn cfDNA được trình tự gen trên hệ thống NextSeq 2000 (Illumina, Mỹ) với độ phủ sâu trung bình 100,000X mỗi amplicon. Các amplicon có độ phủ dưới 10,000X được coi là không đạt chuẩn. Việc phát hiện biến thể được thực hiện sử dụng mpileup từ Samtools như được mô tả trước đây (v1.11) (Nguyen và cộng sự, 2022; Nguyen Hoang và cộng sự, 2023). Mẫu huyết tương có ít nhất một đột biến với VAF trên 0.05% được xác định là dương tính với ctDNA.
Phân tích thống kê
Mức độ tuyến tính giữa kết quả K4C và tiêu chuẩn tham chiếu đã được đánh giá sử dụng các giá trị R2 xuất phát từ phương pháp mô hình tuyến tính “lm” trong gói thống kê R v4.3.0. Tất cả các biểu đồ đại diện được tạo ra bởi phiên bản máy chủ Rstudio 2022.07.02 (Nhóm Rstudio, Mỹ).
Kết quả
Quy trình thử nghiệm
Bảng gen được biên soạn K4C của 473 gen liên quan đến ung thư bao gồm toàn bộ exon, các vùng promoter và intron được chọn, với tổng kích thước bộ gen là 1.7 Mb (Bảng Phụ lục S3). Mẫu FFPE và máu được yêu cầu cho phân tích (Hình 1). DNA nội bào từ khối u và mẫu bạch cầu WBC tương ứng được giải trình tự để xác định các biến thể soma và dòng mầm trong 473 gen. Kết quả này trả lời cho các bác sĩ những thông tin 1) đột biến soma có thuốc điều trị và kháng thuốc giúp dự đoán khả năng đáp ứng với các liệu pháp nhắm mục tiêu đã được FDA chấp thuận; 2) cho biết trạng thái TMB và MSI để quyết định liệu pháp miễn dịch; 3) các biến thể dòng mầm gây bệnh hoặc có khả năng gây bệnh để đánh giá nguy cơ di truyền. Đối với 6 loại ung thư được chọn, tất cả các đột biến xuất phát từ khối u sẽ được xếp hạng bởi thuật toán độc quyền của Gene Solutions và các đột biến tần suất cao nhất sẽ được sử dụng để phát hiện sự hiện diện của ctDNA trong huyết tương bằng cách sử dụng xét nghiệm mPCR riêng biệt. Kết quả này được sử dụng để 4) phát hiện theo dõi tồn dư tối thiểu (MRD) và đánh giá đáp ứng điều trị cho bệnh nhân ung thư.
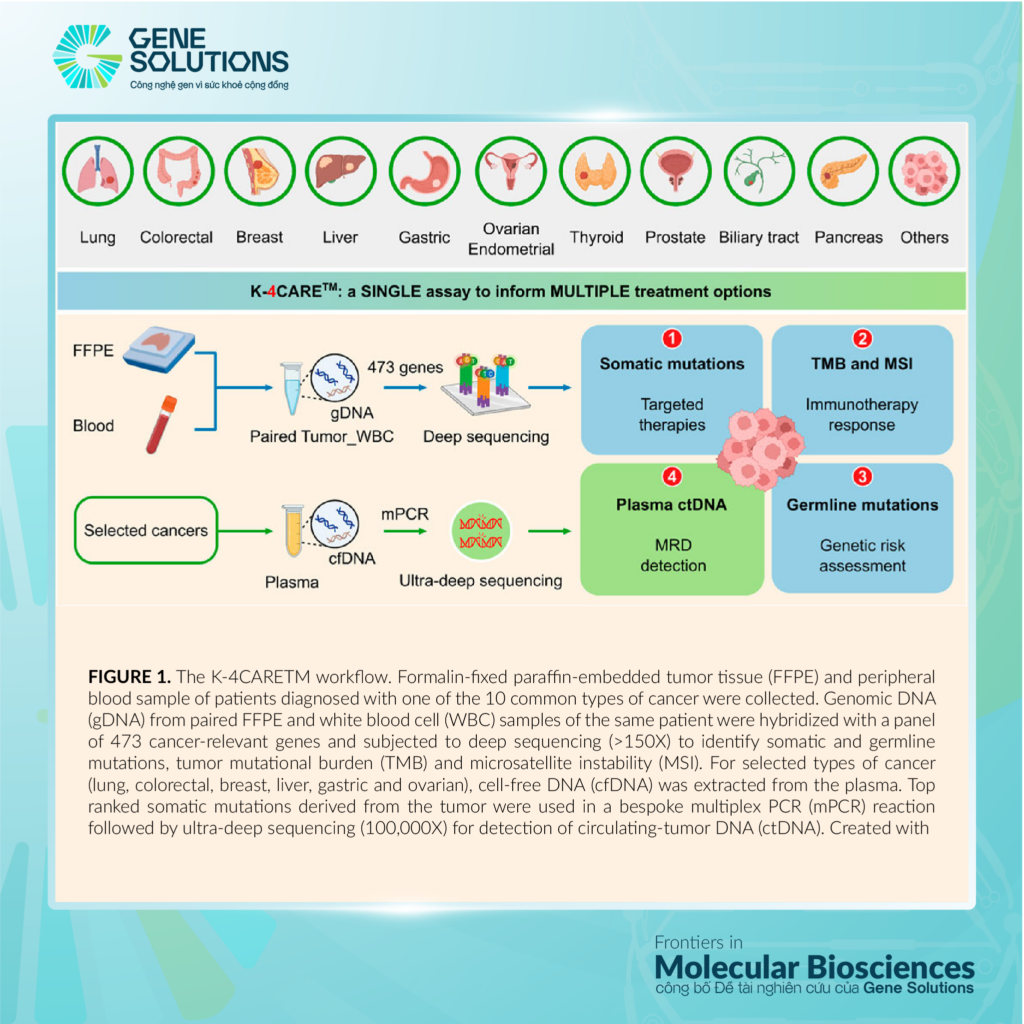
HÌNH 1
Hình 1. Quy trình xét nghiệm K-4CARE™. Mô u cố định bằng formalin và đúc paraffin (FFPE) và mẫu máu ngoại vi của bệnh nhân được chẩn đoán mắc một trong 10 loại ung thư phổ biến được thu thập. DNA genomic (gDNA) từ mẫu FFPE và tế bào bạch cầu (WBC) của cùng một bệnh nhân được lai với một panel 473 gen liên quan đến ung thư và được tiến hành giải trình tự sâu (>150X) để xác định các đột biến somatic và germline, gánh nặng đột biến khối u (TMB) và mất ổn định vi vệ tinh (MSI). Đối với các loại ung thư được chọn (phổi, đại tràng, vú, gan, dạ dày và buồng trứng), DNA tự do (cfDNA) được chiết xuất từ huyết tương. Các đột biến somatic được phát hiện từ tế bào khối u sẽ sử dụng trong phản ứng PCR đa mồi (mPCR), sau đó được giải trình tự với độ phủ sâu (100,000X) để theo dõi DNA khối u tự do lưu hành trong máu (ctDNA). Được tạo ra với BioRender.com.
Đầu tiên, việc lượng giá phân tích được thực hiện để xác định độ chính xác và tính lặp lại của xét nghiệm K4C sử dụng các chuẩn tham chiếu. Sau đó, mẫu lâm sàng từ hơn 10 loại ung thư được sử dụng để đánh giá thêm tính chính xác của xét nghiệm, đặc biệt là khi so sánh với các phương pháp đối chứng khác. Năng lực tổng thể của nghiên cứu K4C được tóm tắt trong Bảng 1.
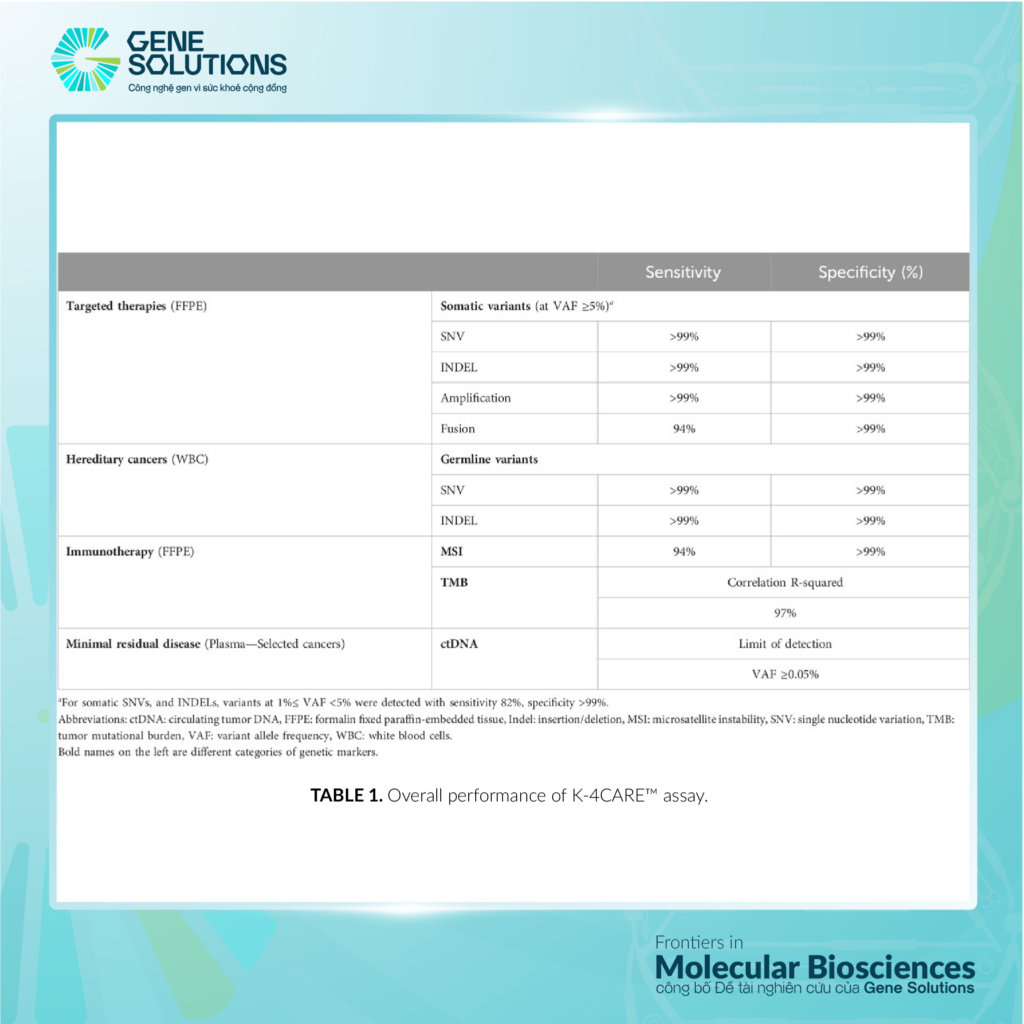
BẢNG 1. Năng lực tổng thể của xét nghiệm K-4CARE™.
Phát hiện biến thể somatic và germline
Theo theo tham chiếu chuẩn là OncoSpan gDNA, có 228 biến thể được xác minh (211 SNV, 17 INDEL) trong panel gen của xét nghiệm K4C của Gene Solutions. Độ nhạy của nghiên cứu K4C ước lượng từ 11 bản sao: hai đến ba bản sao/mỗi lần chạy và chạy 4 lần độc lập. Đối với việc phát hiện SNV và Indel, độ nhạy ước lượng >99% cho VAF ≥5% (Bảng phụ lục S4). Có mối tương quan cao (R2 > 0.90) giữa VAFs dự kiến và VAFs được xác định bởi xét nghiệm K4C (Hình 2A, Bảng phụ lục S5). Độ đặc hiệu >99% cho phát hiện SNV và Indel somatic bằng cách sử dụng Tru-Q 0 (Bảng phụ lục S6). Giới hạn phát hiện (LOD) của xét nghiệm là mức VAF tối thiểu có thể phát hiện được trong ít nhất 95% các bản sao kỹ thuật cùng một nguyên liệu đầu vào. Sử dụng OncoSpan chuẩn, LOD cho SNV và Indels được xác định ở VAF 5% (Bảng phụ lục S7). Các biến thể ở 1% ≤ VAF <5% được phát hiện với độ nhạy 82% và độ đặc hiệu >99% (Bảng phụ lục S6, S7). Độ lặp lại và độ tái lặp của xét nghiệm so sánh với xét nghiệm chuẩn OncoSpan được thể hiện trong Bảng phụ lục S8.
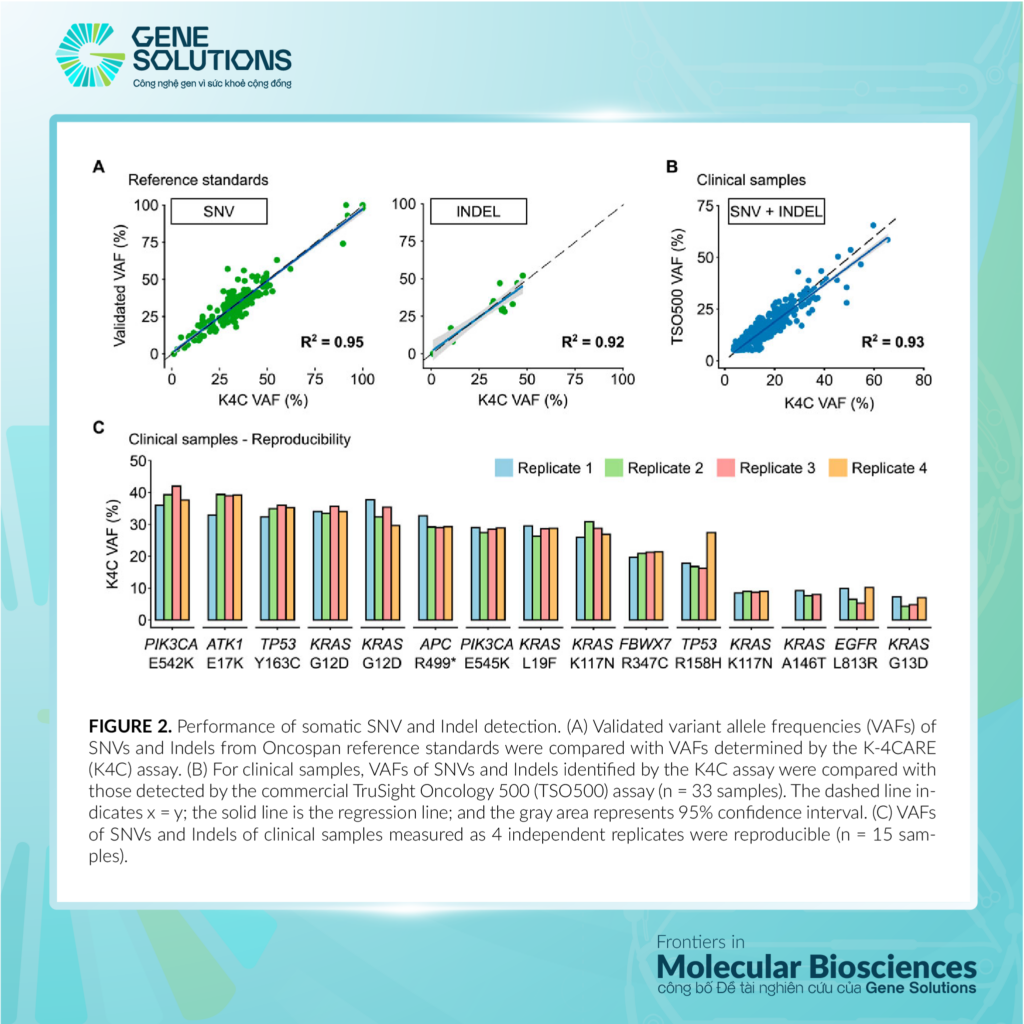
HÌNH 2
HÌNH ẢNH 2. Năng lực phát hiện chuẩn tham chiếu SNV và Indel somatic. (A) Tần số alen đột biến (VAFs) của SNV và Indel từ tiêu chuẩn tham khảo Oncospan được so sánh với VAFs xác định bởi xét nghiệm K-4CARE (K4C). (B) Đối với mẫu lâm sàng, VAFs của SNV và Indel xác định bởi xét nghiệm K4C được so sánh với phát hiện bởi nghiên cứu TruSight Oncology 500 (TSO500) (n = 33 mẫu). Đường nét đứt chỉ ra x = y; đường nét liền là đường hồi quy; và khu vực màu xám thể hiện khoảng tin cậy 95%. (C) VAFs của SNV và Indel của mẫu lâm sàng được đo lường như 4 bản sao độc lập có thể tái sản xuất (n = 15 mẫu).
Tiếp theo, Gene Solutions đã so sánh năng lực phát hiện SNV và Indel của nghiên cứu K4C với TruSight Oncology 500 (TSO500) bằng cách sử dụng 39 mẫu lâm sàng. Sự tương hợp giữa nghiên cứu K4C và TSO500 trong việc phát hiện các đột biến SNV và Indel là 100%. Sự tương quan của VAF được xác định bởi hai nghiên cứu là 0.93 (Hình 2B). Độ tái lặp cao của VAF được đo bởi nghiên cứu K4C cũng được chứng minh trong các mẫu lâm sàng đo trong 4 bản sao độc lập (Hình 2C, Bảng phụ lục S9).
Đối với việc khuếch đại, Gene Solutions sử dụng CNV Mix của vú, phổi và não bao gồm 6 CNV (ERBB2, FGFR3, MYC, EGFR, MET, MYCN) với mỗi 3 bản sao của mỗi loại. Nghiên cứu K4C có thể xác định được sự khuếch đại biến đổi trong tất cả sáu gen, trong khi các tiêu chuẩn tham khảo âm tính không phát hiện được sự khuếch đại nào (Bảng phụ lục S10). Đối với sự hợp nhất gen, tiêu chuẩn hợp nhất của 6 loại ung thư Pan Cancer được sử dụng có chứa sáu loại hợp nhất: TPM3-NTRK1, QKI-NTRK2, ETV6-NTRK3, EML4-ALK, CCDC6-RET và SLC34A2-ROS1. Nghiên cứu K4C phát hiện được 94% sự kiện hợp nhất trong 3 lần tái tạo độc lập, trong khi nó không phát hiện được bất kỳ sự kiện nào trong các tiêu chuẩn tham khảo âm tính (Bảng phụ lục S11).
Đối với các đột biến Germline, nghiên cứu K4C phát hiện được cả biến thể SNV và Indel, BRCA di truyền I, với độ nhạy và độ đặc hiệu lớn hơn 99%; và sự tương quan cao giữa VAF dự kiến và VAF xác định bởi nghiên cứu K4C (Bảng phụ lục S12).
Gánh nặng đột biến của khối u (TMB) nghiên cứu K4C xác định TMB của SeraSeq® TMB-7 và -13 lần lượt là 7.7 và 14.2, nằm trong phạm vi khuyến nghị của nhà sản xuất. Vì các tiêu chuẩn này có tỉ lệ khối u là 100%, điều mà hiếm khi áp dụng cho các mẫu lâm sàng, Gene Solutions đã đánh giá sự ổn định của TMB ở tỉ lệ khối u thấp hơn và hiệu suất giải trình tự khác nhau. Dữ liệu Fastq của tiêu chuẩn khối u được trộn với tiêu chuẩn WBC theo tỉ lệ 3:1, 1:1, 2:3, 3:7, 1:4, và 1:9 để tạo ra các tỉ lệ khối u giả định từ 75% đến 10%; tiếp theo bằng cách lấy mẫu đọc ngẫu nhiên để thay đổi độ sâu trung bình và phần trăm phủ sóng tại 100X trên các tỉ lệ khối u khác nhau. Gene Solutions phát hiện ra rằng giá trị TMB vẫn ổn định khi tỉ lệ khối u ít nhất là 30%, độ sâu trung bình ít nhất là 150X, và phần trăm phủ sóng tại 100X bằng hoặc vượt quá 75% (Hình 3A).
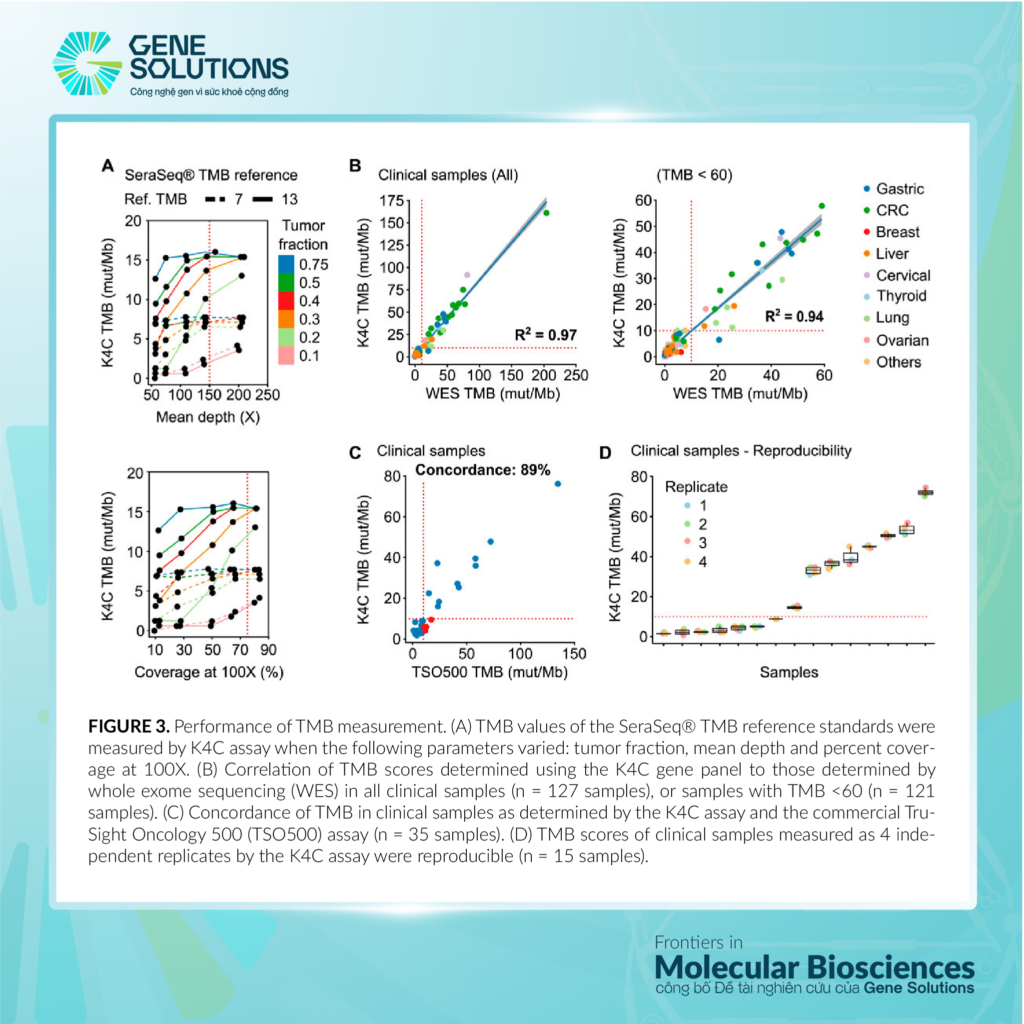
HÌNH 3
HÌNH 3. Năng lực đo lường TMB. (A) Các giá trị TMB của các tiêu chuẩn tham khảo TMB SeraSeq® được đo bởi nghiên cứu K4C khi các tham số sau đây thay đổi: tỉ lệ khối u, độ sâu trung bình và phần trăm phủ sóng tại 100X. (B) Sự tương quan của các điểm số TMB được xác định bằng panel gen K4C so với những điểm số được xác định bằng giải trình tự toàn bộ exome (WES) trong tất cả các mẫu lâm sàng (n = 127 mẫu), hoặc các mẫu có TMB <60 (n = 121 mẫu). (C) Sự phù hợp của TMB trong các mẫu lâm sàng được xác định bởi nghiên cứu K4C và nghiên cứu TruSight Oncology 500 (TSO500) (n = 35 mẫu). (D) Các điểm số TMB của các mẫu lâm sàng được đo là 4 lần tái tạo độc lập bởi nghiên cứu K4C có thể tái sản xuất (n = 15 mẫu).
Gene Solutions đã xác định TMB của 127 mẫu lâm sàng sử dụng panel chọn lọc K4C hoặc WES, là tiêu chuẩn vàng cho việc đo lường TMB. Hệ số tương quan của giá trị TMB giữa 2 phương pháp là 0.97 cho tất cả các mẫu lâm sàng, và 0.94 cho các mẫu có TMB <60 (Hình 3B, Bảng phụ lục S13). Khi đánh giá so với nghiên cứu TSO500 sử dụng 39 mẫu lâm sàng, sự phù hợp để xác định tình trạng TMB của nghiên cứu K4C là 89% (Hình 3C, Bảng phụ lục S14). Chỉ số TMB có thể tái sản xuất trong các mẫu lâm sàng được phân tích trong 4 lần chạy độc lập (Hình 3D).
Mất ổn định vi vệ tinh (MSI)
Tình trạng MSI đầu tiên được xác định cho 158 trường hợp ung thư đại trực tràng và dạ dày, sử dụng PCR và phân tích đoạn cho 5 dấu hiệu microsatellite được Khuyến nghị bởi Viện Ung thư Quốc gia (Boland và cộng sự, 1998). Nghiên cứu K4C thực hiện cho 31 mẫu được chọn ngẫu nhiên, bao gồm cả mẫu MSI-H và MSS. Sự phù hợp của nghiên cứu K4C để xác định tình trạng MSI là 97% so với phương pháp PCR (Hình 4A, Bảng phụ lục S15). Khi đánh giá so với nghiên cứu TSO500 sử dụng 39 mẫu lâm sàng, sự phù hợp giữa K4C và TSO500 cho việc đo lường MSI là 98% (Hình 4B, Bảng phụ lục S14). Chỉ số MSI được xác định bởi nghiên cứu K4C rất có thể tái sản xuất trong các mẫu lâm sàng được phân tích trong 4 lần chạy độc lập (Hình 4C).
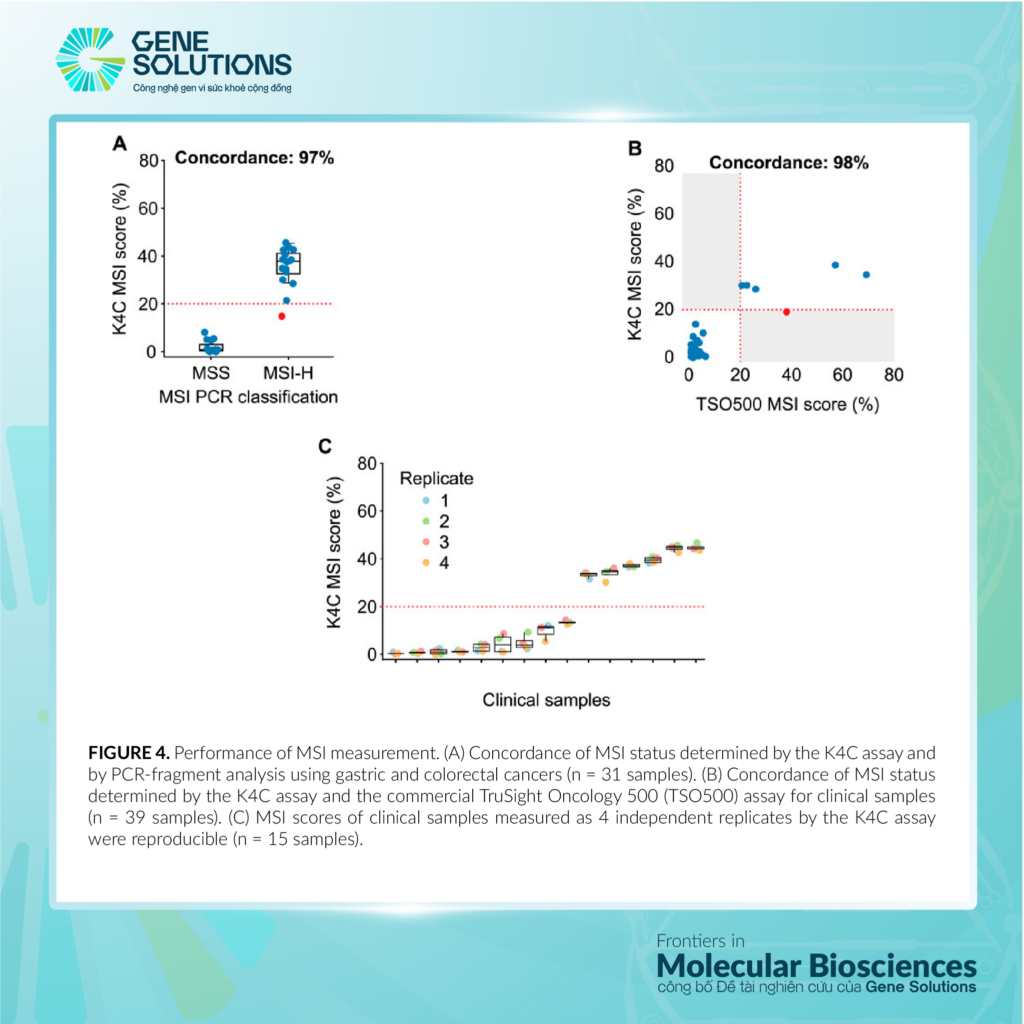
HÌNH 4
HÌNH 4. Năng lực đo lường MSI. (A) Sự phù hợp của tình trạng MSI được xác định bởi nghiên cứu K4C và bằng phân tích PCR-fragment trong ung thư dạ dày và đại trực tràng (n = 31 mẫu). (B) Sự phù hợp của MSI được xác định bởi nghiên cứu K4C và nghiên cứu TruSight Oncology 500 (TSO500) cho các mẫu lâm sàng (n = 39 mẫu). (C) Chỉ số MSI của các mẫu lâm sàng được đo là 4 lần tái tạo độc lập bởi nghiên cứu K4C có thể tái sản xuất (n = 15 mẫu).
Ứng dụng lâm sàng
Để minh họa tính hữu ích lâm sàng của nghiên cứu K4C, Gene Solutions đã xác định đột biến có dự đoán phản ứng điều trị với các thuốc được FDA phê duyệt (chỉ cấp độ 1 và 2 của OncoKB). Phần trăm các mẫu có ít nhất 1 đột biến có thể sử dụng là 68.4%, 57.1%, 50%, 47.1%, 9.1% và 7.1% ở các ung thư tuyến giáp, vú, phổi, CRC, buồng trứng và cổ tử cung tương ứng (Hình 5A, Bảng phụ lục S16). Đánh giá lâm sàng TMB cho thấy chỉ số TMB cao nhất ở ung thư CRC, dạ dày và phổi (Hình 5B). Đối với MSI, tỉ lệ phần trăm của MSI-H là 16.9%, 9.1%, 5.3% và 3.4% ở ung thư CRC, buồng trứng, tuyến giáp và dạ dày (Hình 5C). Gene Solutions cũng đã xem xét mối quan hệ giữa TMB và MSI trong các mẫu lâm sàng (Bảng phụ lục S17). Phần lớn các mẫu ung thư CRC và dạ dày có cả TMB-H và MSI-H. Một số mẫu ung thư phổi và gan có TMB-H nhưng nằm trong trong MSS (Hình 5D).
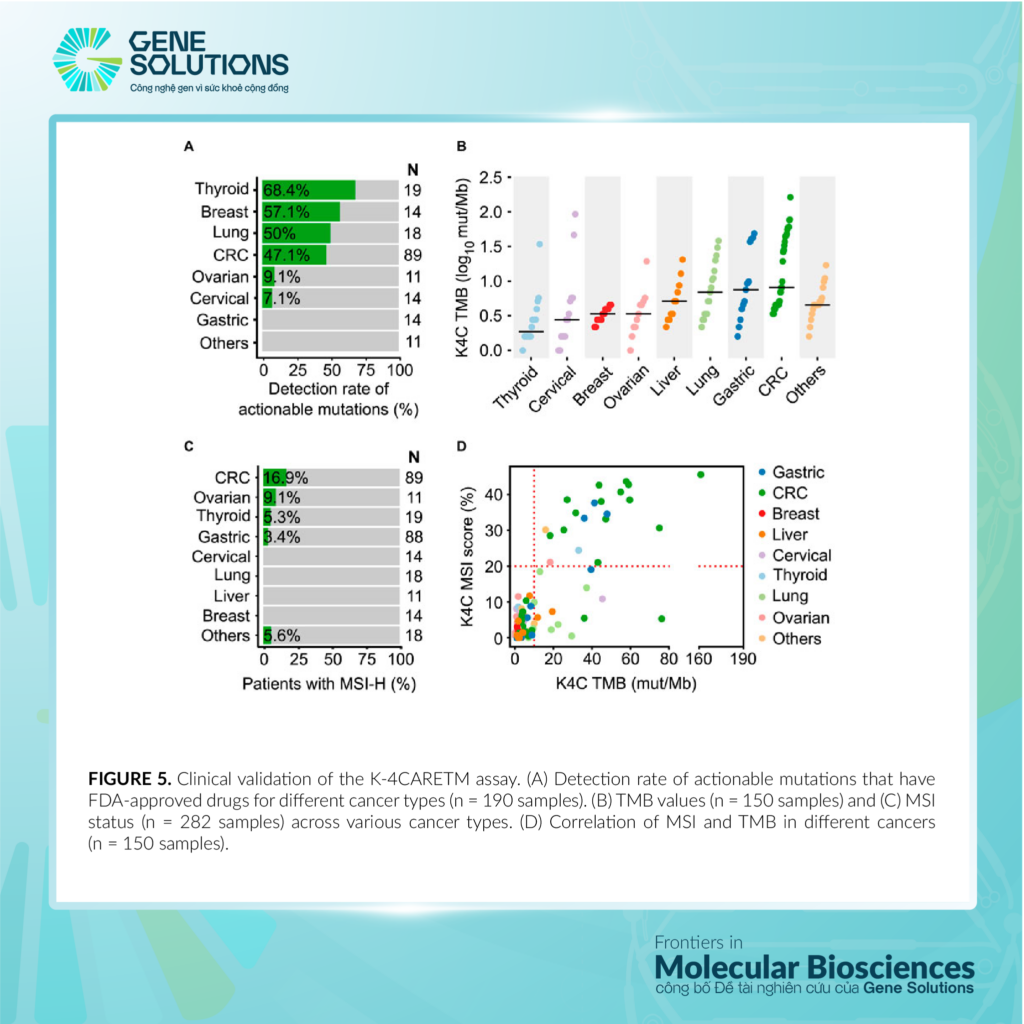
HÌNH 5
HÌNH 5. Đánh giá ứng dụng lâm sàng của nghiên cứu K-4CARE™. (A) Tỷ lệ phát hiện các đột biến có thể sử dụng thuốc được FDA phê duyệt cho các loại ung thư khác nhau (n = 190 mẫu). (B) Các giá trị TMB (n = 150 mẫu) và (C) Tình trạng MSI (n = 282 mẫu) trên các loại ung thư khác nhau. (D) Sự tương quan giữa MSI và TMB trong các loại ung thư khác nhau (n = 150 mẫu).
Phát hiện ctDNA trong huyết tương
Trong 40 bệnh nhân mắc bệnh ung thư đại trực tràng (CRC) hoặc ung thư vú, tổng số các đột biến đặc hiệu khối u được phát hiện là 4257 biến thể. Để sử dụng thuật toán, Gene Solutions đã chọn từ hai đến năm biến thể độc đáo cho mỗi bệnh nhân và tăng cường chúng trong các xét nghiệm mPCR riêng biệt để phát hiện ctDNA trong huyết tương. Trong tổng số 127 biến thể chọn từ các khối u, 100 biến thể (78.7%) được phát hiện trong mẫu huyết tương (Hình 6A). Không có sự tương quan giữa VAF của một biến thể trong mô FFPE và VAF hoặc tình trạng phát hiện trong huyết tương (Hình 6B). Khi cùng một nhóm đột biến được sử dụng để theo dõi ctDNA trong các mẫu huyết tương lâu dài, Gene Solutions có thể phát hiện MRD và tái phát phân tử trước khi chẩn đoán lâm sàng. Hai nghiên cứu trường hợp được trình bày để minh họa tính hữu ích lâm sàng của xét nghiệm. Đầu tiên, bệnh nhân K4C012 được chẩn đoán ung thư vú giai đoạn I, không có hormone. ctDNA được phát hiện trong huyết tương trước phẫu thuật, được loại bỏ sau phẫu thuật nhưng trở lại dương tính sau điều trị hóa chất xạ trị bổ trợ (CRT). Khám lâm sàng chỉ xác nhận tái phát ung thư 10 tháng sau khi ctDNA được phát hiện dương tính (Hình 6C). Thứ hai, bệnh nhân K4C077 được chẩn đoán ung thư đại trực tràng T4aN2M0. Sau phẫu thuật, VAF của ctDNA giảm mạnh nhưng bệnh nhân vẫn dương tính với ctDNA, cho thấy sự hiện diện của tế bào ung thư còn sót lại. Mặc dù đã trải qua CRT, nhưng 10.5 tháng sau, bệnh nhân được chẩn đoán di căn gan. Mức độ của antigen carcinoembryonic (CEA), biomaker được sử dụng để theo dõi tình trạng tiến triển bệnh nhân CRC, kết quả cho thấy các chỉ số vẫn bình thường cả trước và sau phẫu thuật, chỉ tăng lên sau 6 tháng kể từ khi ctDNA được phát hiện dương tính (Hình 6D).
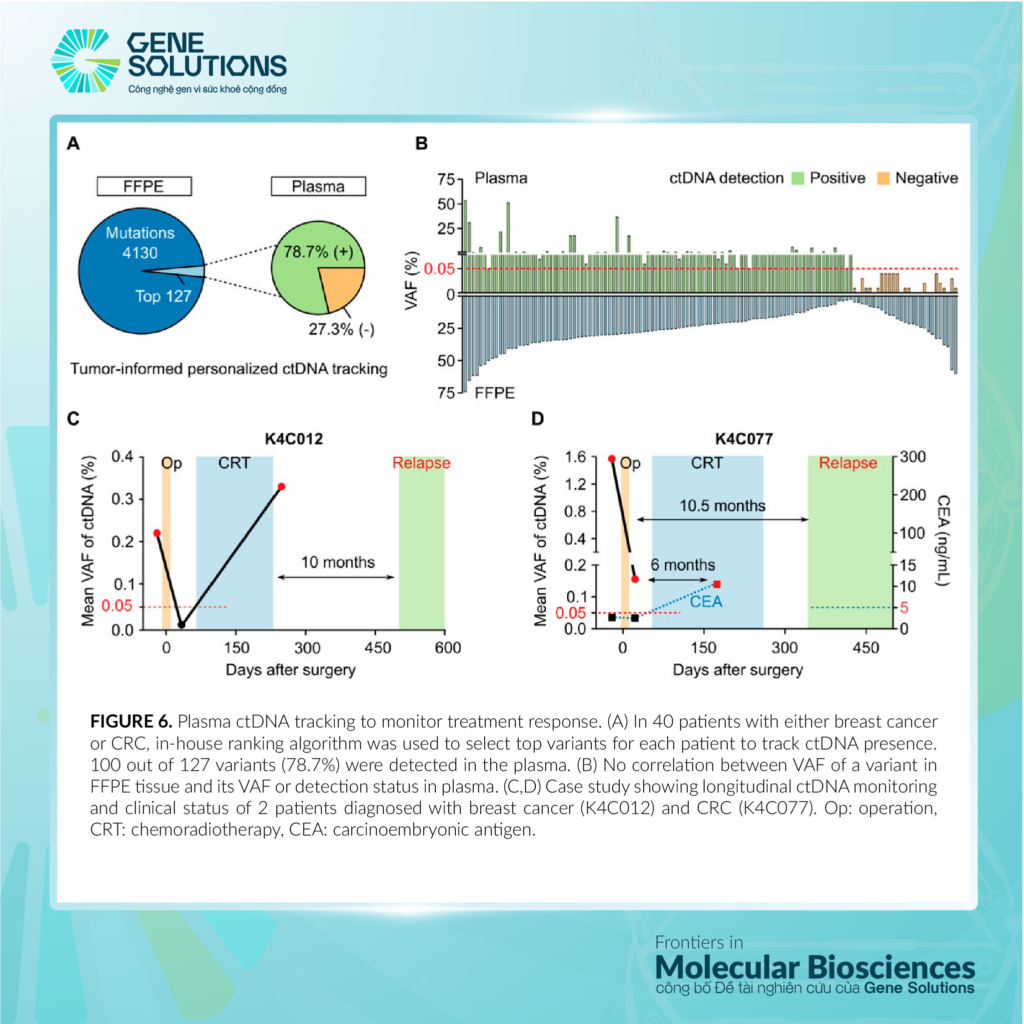
HÌNH 6
HÌNH 6. Theo dõi ctDNA trong huyết tương để giám sát phản ứng điều trị. (A) Trong 40 bệnh nhân mắc bệnh ung thư vú hoặc CRC, thuật toán của Gene Solutions được sử dụng để theo dõi sự hiện diện của ctDNA. 100 trên 127 biến thể (78.7%) được phát hiện trong huyết tương. (B) Không có sự tương quan giữa VAF của biến thể trong mô FFPE và VAF hoặc phát hiện trong huyết tương. (C,D) Nghiên cứu trường hợp cho thấy việc theo dõi ctDNA theo thời gian và tình trạng lâm sàng của 2 bệnh nhân được chẩn đoán mắc bệnh ung thư vú (K4C012) và CRC (K4C077). Op: phẫu thuật, CRT: hóa xạ trị, CEA: antigen carcinoembryonic.
Thảo luận
Trong nghiên cứu này, Gene Solutions đã trình bày lượng giá xét nghiệm và đánh giá ứng dụng lâm sàng của K-4CARE – xét nghiệm CGP dựa trên khối u cho phép phát hiện các đột biến somatic, đột biến germline, đo lường TMB và MSI, cũng như theo dõi ctDNA, tất cả đều thực hiện trong một nền tảng duy nhất. Các chỉ số năng lực của nghiên cứu đã được thiết lập và đánh giá nghiêm ngặt, sử dụng cả tiêu chuẩn tham chiếu và các phương pháp đối chứng.
Đầu tiên, nghiên cứu K4C có thể phát hiện 4 loại đột biến somatic với độ nhạy và độ đặc hiệu cao, chỉ sử dụng DNA khối u. Không giống như các xét nghiệm CGP khác sử dụng RNA để phát hiện sự tái sắp xếp của gen (White et al., 2021; Ng et al., 2022; Saldivar et al., 2023), Gene Solutions đã thiết kế các đầu dò nhắm vào các khu vực intron của các gen thường xuyên hợp nhất, và các thư viện DNA được sử dụng để kết hợp với các đầu dò này. Đường ống bioinformatic được sử dụng để phát hiện sự hợp nhất gen tương tự như những nghiên cứu trước đây (Wang et al., 2023; Xia et al., 2023; Ye et al., 2023). Vì điều này loại bỏ việc xử lý RNA khối u, nó giúp tăng tốc kết quả và làm cho nghiên cứu hiệu quả hơn về chi phí và tính khả thi, đặc biệt là cho các nước đang phát triển như Việt Nam – nơi chất lượng RNA thu được từ mẫu FFPE thường không đủ cho NGS (trích theo dữ liệu từ thông tin nội bộ). Hơn nữa, Gene Solutions đã chứng minh tính hữu ích lâm sàng của nghiên cứu để xác định ít nhất 1 đột biến có thể sử dụng (cấp độ 1–2 của OncoKB) trong 38.9% các mẫu lâm sàng (74/190). Tỷ lệ này dường như cao hơn so với các nghiên cứu CGP trong các nhóm khác, ví dụ như 12.0% trong Ng et al (Ng et al., 2022), 10.7% trong Aoyagi et al (Aoyagi et al., 2022) và 23.7% trong Karol et al (Karol et al., 2021). Có thể do phân bố mẫu qua các loại ung thư khác nhau trong mỗi nhóm. Phân tích của Gene Solutions bao gồm ung thư CRC (46.8%) và ung thư tuyến giáp (10.0%), so với các loại ung thư khác thì cả hai đều có tỉ lệ cao về động biến có thể có thể sử dụng. Đặc biệt đối với ung thư tuyến giáp thể nhú (papillary), Gene Solutions đã xác định tỉ lệ phần trăm của các đột biến BRAF V600E trong 68.4% các mẫu, nằm ở phạm vi cao được báo cáo (32%–65%) (Xing et al., 2013). Vì kích thước mẫu trong từng loại ung thư khá nhỏ, do đó kết quả này chỉ được sử dụng để minh họa cho ứng dụng lâm sàng của nghiên cứu, chứ không cung cấp đột biến đại diện hay mức độ cụ thể có thể thực hiện thực sự. Phân tích toàn diện hơn về hồ sơ đột biến có thể hành động trong các nhóm lớn hơn nhiều về ung thư phổi, ung thư vú và ung thư đại trực tràng đã được báo cáo trong các nghiên cứu trước đây của Gene Solutions (Dang et al., 2020; Nguyen et al., 2022; Nguyen Hoang et al., 2023).
Đối với việc đo lường TMB, Gene Solutions đã bao gồm các đột biến không đồng nghĩa trong một vùng genomic khoảng 1.7 Mb, lớn hơn so với 1 Mb được khuyến nghị để đảm bảo độ chính xác và ổn định của TMB (Buttner et al., 2019; Bevins et al., 2020). Quy trình xác thực đã được căn chỉnh với các tiêu chuẩn hiện tại và các xét nghiệm CGP trước đó (Merino et al., 2020; White et al., 2021; Ng et al., 2022; Saldivar et al., 2023), cho thấy sự tương quan cao giữa bảng K4C và tiêu chuẩn vàng WES (R2 = 0.97). Sự phù hợp của việc đo lường TMB giữa K4C (khối u-bình thường) và TSO500 (chỉ khối u) là 89%, thấp hơn một chút so với sự phù hợp giữa các bảng chỉ khối u (>90%) (Bevins et al., 2020). Gene Solutions nhận thấy rằng tất cả các mẫu không phù hợp đều có TMB TSO500 cao hơn so với giá trị K4C, điều này cũng được báo cáo trong một nghiên cứu CGP tumor-normal khác (Saldivar et al., 2023). Sự khác biệt như vậy có thể do không có mẫu bình thường tương ứng và việc bao gồm các đột biến đồng nghĩa trong thuật toán tính toán TMB của nghiên cứu TSO500 (Saldivar et al., 2023). Ngoài ra, trong số các mẫu lâm sàng của Gene Solutions, các loại ung thư có Chỉ số TMB cao nhất là CRC, ung thư dạ dày và ung thư phổi. Kết quả này phù hợp với các nghiên cứu trước đó (Lawrence et al., 2013; Wu et al., 2022; Saldivar et al., 2023) nhưng khác biệt so với nghiên cứu của Ng et al báo cáo TMB cao nhất ở ung thư gan và ung thư vú trong một nhóm người Châu Á (Ng et al., 2022).
Đối với phân tích MSI, nghiên cứu dựa trên IHC và PCR khảo sát 5 dấu hiệu lặp lại mononucleotide vẫn là lựa chọn đầu tiên trong các ung thư thuộc phổ của hội chứng Lynch như ung thư đại trực tràng (CRC) và ung thư nội mạc tử cung (Luchini et al., 2019). Đánh giá MSI bằng NGS cũng được khuyến nghị là một lựa chọn cho các ung thư này để có thêm lợi ích của việc khảo sát TMB, một dấu ấn sinh học khác cho ICIs (Luchini et al., 2019). Đối với tất cả các ung thư không liên quan đến hội chứng Lynch, NGS là phương pháp lựa chọn để xác định MSI vì độ tin cậy của IHC và 5 dấu hiệu bằng PCR chưa được chứng minh (Luchini et al., 2019). Trong phân tích của Gene Solutions sử dụng mẫu ung thư CRC và dạ dày, Gene Solutions đã chứng minh sự phù hợp của MSI đo bằng PCR cho 5 dấu hiệu lặp lại và NGS khảo sát khoảng 800 vị trí lặp lại, cho thấy độ tin cậy và chính xác của kỹ thuật NGS. Tỉ lệ phần trăm của MSI-H là 16.9% trong CRC, con số tương tự như tỉ lệ đã được báo cáo trước đây (Bonneville et al., 2017; Middha et al., 2017). Tỉ lệ phần trăm của MSI-H trong mẫu ung thư dạ dày (3.4%) thấp hơn so với Bonneville et al (19.9%) (Bonneville et al., 2017) nhưng tương đương với các nhóm lớn khác (2.5%–3.4%) (Middha et al., 2017; Trabucco et al., 2019). Tỉ lệ cao bất ngờ của MSI-H trong ung thư buồng trứng và ung thư tuyến giáp trong nhóm của Gene Solutions cần được điều tra thêm với kích thước mẫu lớn hơn.
Kể từ khi PD-L1, MSI và TMB đã được phê duyệt là các dấu ấn sinh học độc lập cho ICIs, mối quan hệ giữa ba dấu ấn sinh học này đã được khám phá rộng rãi trong các nghiên cứu trước đây. PD-L1 được tìm thấy có sự phù hợp thấp với TMB và MSI, trong khi mối quan hệ giữa TMB và MSI thay đổi đáng kể theo loại ung thư (Luchini et al., 2019; Wu et al., 2022). Sự phù hợp cao nhất của TMB và MSI được tìm thấy trong ung thư đại trực tràng và ung thư dạ dày, và gần như không có trong ung thư phổi (Luchini et al., 2019; Wu et al., 2022). Điều này hoàn toàn phù hợp với quan sát của Gene Solutions trong nghiên cứu. Hơn nữa, tương tự như những nghiên cứu, Gene Solutions cũng tìm thấy TMB-H có thể xảy ra trong các mẫu MSS qua các loại ung thư khác nhau, trong khi sự xuất hiện của MSI-H ở các mẫu TMB-L là khá hiếm. Điều này một lần nữa nhấn mạnh tầm quan trọng của việc kiểm tra toàn diện cho nhiều dấu ấn sinh học để tối đa hóa cơ hội điều trị cho bệnh nhân. Cũng cần lưu ý rằng việc giải thích các dấu ấn sinh học này phải cụ thể theo loại ung thư, vì ngưỡng cho TMB-H ở 10 mut/Mb, ví dụ, chỉ được chứng minh là áp dụng cho ung thư phổi (Samstein et al., 2019).
Cuối cùng, một ứng dụng độc đáo của nghiên cứu K4C là khả năng sử dụng các biến thể cá nhân hóa được phát sinh từ khối u để theo dõi sự hiện diện của ctDNA trong huyết tương. Sự xác thực phân tích của kỹ thuật mPCR đã được làm rõ trong các nghiên cứu trước đây của Gene Solutions (Nguyen et al., 2022; Nguyen Hoang et al., 2023). Tại giới hạn phát hiện thấp nhất là 0.05%, nghiên cứu đã xác định chính xác ctDNA trong huyết tương sử dụng các đột biến được xếp hạng bởi thuật toán độc quyền của Gene Solutions. Giá trị tiên lượng và dự đoán của ctDNA trong việc theo dõi thường xuyên bệnh nhân ung thư trong và sau điều trị đã được chứng minh một cách nhất quán trong nhiều thử nghiệm lâm sàng (Chin et al., 2019). Cụ thể đối với ICIs, việc loại bỏ ctDNA sau 2 chu kỳ điều trị đã được chứng minh là một chỉ báo mạnh mẽ của phản ứng lâu dài trong nhiều khối u rắn (Cabel et al., 2017; Bratman et al., 2020).
Tổng hợp lại, dữ liệu được trình bày trong nghiên cứu này nhấn mạnh độ chính xác và độ tin cậy của nghiên cứu K4C trong việc cung cấp các biến đổi và dấu hiệu di truyền toàn diện để thông tin cho các liệu pháp nhắm trúng đích và miễn dịch cho bệnh nhân ung thư. Nghiên cứu cũng có thể phát hiện ctDNA trong huyết tương, sau đó có thể được sử dụng để theo dõi phản ứng điều trị một cách thường xuyên và phát hiện ung thư còn sót lại.
Công bố dữ liệu có sẵn công khai
Các bộ dữ liệu được trình bày trong nghiên cứu này có thể được tìm thấy trong các kho lưu trữ trực tuyến. Tên của kho lưu trữ và số(s) mã truy cập có thể được tìm thấy dưới đây: https://www.ncbi.nlm.nih.gov/bioproject/PRJNA1035299/
Tuyên bố đạo đức
Các nghiên cứu liên quan đến con người đã được phê duyệt bởi ủy ban đạo đức của Đại học Y Dược, Thành phố Hồ Chí Minh (số phê duyệt 164/HDDD). Sử dụng mẫu huyết tương cho việc theo dõi ctDNA được thực hiện dưới sự phê duyệt của ủy ban đạo đức của Đại học Y Dược, Thành phố Hồ Chí Minh (số phê duyệt 14/GCN-HDDD cho ung thư đại trực tràng, 300/HDDD cho ung thư vú). Các nghiên cứu đều được tiến hành phù hợp với pháp luật và yêu cầu của cơ sở. Đồng thời, đề tài cũng đã được sự đồng ý và chấp thuận của những người tham gia nghiên cứu.
Đóng góp của tác giả
T-PN: Viết bản thảo gốc, Biên tập và đánh giá bản thảo, Khái niệm hóa, Phân tích chính thức, Điều tra, Phương pháp luận, Trực quan hóa. TN: Quản lý dữ liệu, Phân tích chính thức, Phương pháp luận, Phần mềm, Biên tập và đánh giá bản thảo. NT: Quản lý dữ liệu, Trực quan hóa, Biên tập và đánh giá bản thảo, Phân tích chính thức, Phương pháp luận, Viết bản thảo gốc. V-AN: Quản lý dữ liệu, Trực quan hóa, Biên tập và đánh giá bản thảo. HT: Quản lý dữ liệu, Biên tập và đánh giá bản thảo. V-UT: Quản lý dữ liệu, Biên tập và đánh giá bản thảo. YN: Quản lý dữ liệu, Biên tập và đánh giá bản thảo. AN: Quản lý dữ liệu, Biên tập và đánh giá bản thảo. CN: Quản lý dữ liệu, Biên tập và đánh giá bản thảo. TD: Phân tích chính thức, Biên tập và đánh giá bản thảo. DN: Phân tích chính thức, Biên tập và đánh giá bản thảo. H-NN: Phân tích chính thức, Biên tập và đánh giá bản thảo, Giám sát. HG: Phân tích chính thức, Phần mềm, Biên tập và đánh giá bản thảo, Giám sát. LT: Khái niệm hóa, Phân tích chính thức, Điều tra, Phương pháp luận, Quản lý dự án, Giám sát, Viết bản thảo gốc, Biên tập và đánh giá bản thảo.
Tài trợ
Tác giả tuyên bố đã nhận được sự hỗ trợ tài chính cho nghiên cứu, tác giả, và/hoặc việc xuất bản bài viết này. Nghiên cứu này được tài trợ bởi Gene Solutions, Việt Nam. Nhà tài trợ không có vai trò nào trong thiết kế nghiên cứu, thu thập và phân tích dữ liệu, hoặc chuẩn bị bản thảo.
Lời cảm ơn
Chúng tôi cảm ơn Hoa Phan, Viện Di truyền Y học, Việt Nam đã hỗ trợ trong việc chuẩn bị bản thảo.
Xung đột lợi ích
Các tác giả T-PN, TN, NT, V-AN, AN, HT, V-UT, YN, AN, CN, DN, H-NN, HG và LT đều là nhân viên của Gene Solutions.
Tác giả công bố rằng nghiên cứu được thực hiện không mang tính thương mại hay yếu tố tài chính nào có thể được coi là xung đột lợi ích tiềm ẩn.
Lưu ý của nhà xuất bản
Tất cả các công bố được thể hiện trong bài viết này chỉ thuộc về các tác giả và không phản ánh quan điểm của các tổ chức liên quan, hoặc của nhà xuất bản, biên tập viên và người đánh giá. Bất kỳ sản phẩm được đánh giá trong bài viết này, hoặc tuyên bố không được đảm bảo hoặc chứng thực bởi nhà xuất bản.
Tài liệu tham khảo
Xem thêm thông tin về công trình nghiên cứu tại: Frontiers in Molecular Biosciences